论文地址:Med3D: Transfer Learning for 3D Medical Image Analysis
在该论文中,已经被训练好了的通用的编码器才是最核心的,可以用于处理其他图像任务。
1.文章亮点
- 我们的目标是建立一个通用的三维骨干网,可以转移到其他医疗任务,获得比从零开始训练更好的性能。
- 为了解决当前医学3D图像数量少,标注困难的问题,腾讯优图提出了用大量的医学3D图像训练出一个预训练的模型,有利于用迁移学习加速其他具体的图像任务的收敛和提高精度。
- 当前的一些方法并不能很好地利用3D医学图图像的空间三维信息。由于缺乏大规模的三维医学图像,基于多域三维医学图像数据集的协同训练模型可能是一种解决方案。
- 主要内容:建立一个大型的三维医学数据集(内含多种种类的分割问题),并利用这些数据训练一个基线网络,这些基线网络可以用来解决其他医学问题。
- 针对不完全标注问题,提出了一种多分支译码器(A multi-branch decoder)
2.论文的贡献
- 提出了一种针对三维多域医学数据的异构Med3D网络,它可以在数据域分布差异较大的情况下提取一般的三维特征。
- 将Med3D模型的主干转移到三个新的三维医学图像任务中。并通过大量实验验证了Med3D的有效性和有效性。
- 为了方便社区复制实验结果并将Med3D应用于其他应用,Med3D预训练模型和相关源代码将开源。
3 方法
3.1 数据收集
数据集来自各个不同的3D医学图像分割数据集以得到更好的、更具代表性的信息,而不是分类数据集。 原因:
- ①与自然景象中的图像相比,医学图像中含有的物体类别较少,物体类别更少更容易导致网络的泛化能力差。
- ②与自然景象中的图像相比,在3D医学图像中分类标签是一个很弱的监督信息。因为分类标签可能只与图像中很小的一部分有关联,这不利于网络的收敛。
3.2 归一化
实验中所用的数据集的形态(MRI、CT)、三维空间分辨率和像素强度范围都不相同,有必要进行空间分辨率和灰度值的分布归一化。
- 空间(分辨率)归一化Spatial Normalization:由于不同的设备,来自不同医院或中心的图像在空间距离中存在差异,这种距离指的是在不同的领域中,两个像素之间的物理距离是不同的。而且这种物理信息是不可能被CNN所学习的。Spatial Normalization即重采样到相同的分辨率,以减少立体空间像素变化的影响。(处理过程尚未明白,待补充)
- 像素强度(灰度值)归一化:(对每张图片)svd标准差,vm均值,vi原始值
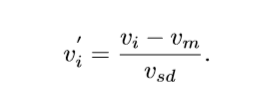
3.2 Med3D Network
采用了通用的编码解码分割结构,其中编码器可以是任何基本结构。在这项工作中,采用了ResNet模型家族作为编码器的基本结构,并做了一些小的修改,使网络能够训练三维医疗数据。数据集的缺点:缺少多器官分割注释即不完全标注问题。例如:肝分割数据集中只有肝的作为前景,其他的器官等都被当做背景,其他数据集也类似。这种不完整的标注信息会使网络产生混淆,训练过程不收敛。
由于在大规模的三维医学数据中对完整的器官图谱进行详细的注释在技术上是不可能的,针对不完全标注问题,提出了一种多分支译码器。
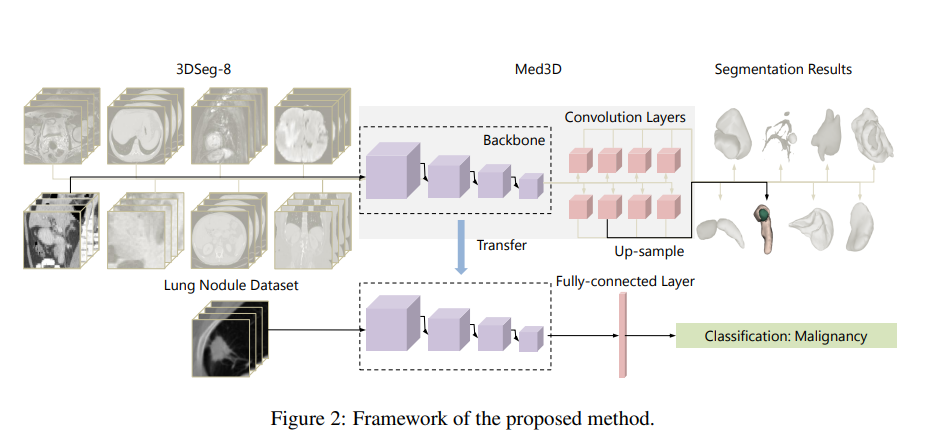
- 用八个特定的解码器分支连接编码器,其中每一个都对应于3DSeg-8(八个不同分割数据集)的一个特定数据集。
- 在训练阶段,每个解码器分支只处理从对应数据集中提取的特征图,其余分支不参与其优化过程。此外,每个解码器分支共享相同的架构,即使用一个卷积层来组合来自编码器的特性。(即使用相同的编码器,但是使用不同解码器)
- 计算网络输出与地面真值标注之间的差异:直接从解码器上采样特征映射到原始图像大小。这种简单的译码器设计使网络能够集中精力训练一名通用编码器。在测试阶段,解码器部分被移除,剩下的编码器可以转移到其他任务。
4.举例
4.1肺分割
-先将编码部分从Med3D中提取出来作为特征提取部分,然后对整个身体的肺进行分割,然后是三组3D解码层。 三组解码层:
- 第一组解码器:核大小为(3,3,3)、通道数256(用于将feature map放大两倍)的转置卷积层和(3,3,3)内核大小、128个通道的卷积层构成。
- 第二组解码器:剩下的两组解码器层与第一组解码器层相似,每层解码器层的信道数逐渐增加一倍。
- 最后,利用(1,1,1)核中的卷积层生成最终输出,信道数对应于类别数
4.2肝分割
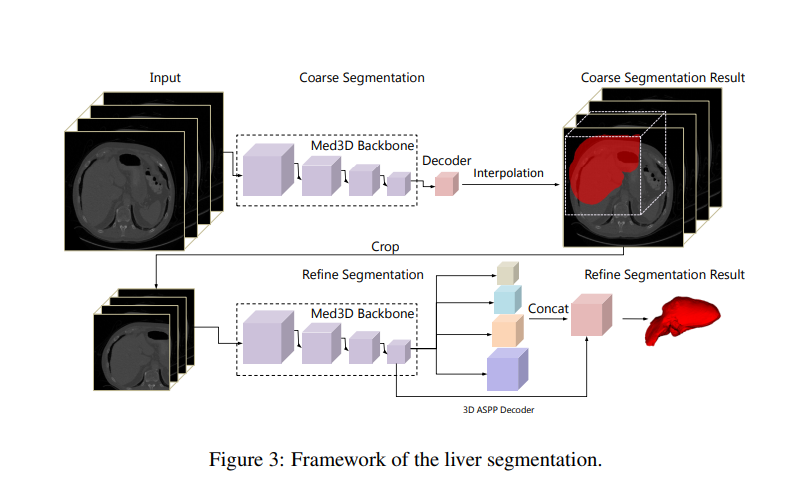
受2018年三维心房分割大赛[34]第一名的启发,我们采用两阶段分割网络对肝脏进行分割。
分割框架图:
-
首先,在整个图像中对肝脏进行粗略分割,得到目标的感兴趣区域(ROI)。
在此阶段: ①我们将Med3D预训练的骨干(backbone pre-trained from Med3D)作为编码器部分进行传输。然后以核为(1,1,1)、通道数为2(肝脏与背景)的卷积层。 ②对图像进行大小调整后,将图像输入粗分割网络进行32次下采样提取特征。 ③然后,用双线性插值方法将特征图上采样到原始图像的大小。
-
然后,根据第一阶段的结果对肝脏靶区进行裁剪,再对靶区进行细分,得到最终的肝脏分割结果。这一阶段主要是肝脏轮廓的精细分割。
为了在特征图中获得更密集的尺度信息,获得更大的接受域,我们嵌入骨干预培训(backbone pre-trained from Med3D)从Med3D到最先进的DenseASPP分割网络,该网络以密集的方式连接一组空洞卷积层,生成的多尺度特征不仅覆盖更大的尺度范围,而且在不显著增加模型大小的情况下密集覆盖该尺度范围。然后,我们用相应的3D版本替换所有的2D内核。 由于第一步的结果不可避免地存在ground truth与liver目标预测之间的偏差,为了提高精细分割模型的鲁棒性,我们随机扩张肝脏靶区,然后用旋转和平移两种方法对其进行处理。
5 实验
5.1 Transfer learning实验
将预先训练好的Med3D编码器与DenseASPP[35]网络连接起来,并使用一个模型演示了在肝脏分割任务上的最新性能。建立了与ResNet-152骨干网相同的分割网络,并用预先训练好的Med3D对其进行初始化。 在训练过程中,由于某些卷没有给出间距信息,我们用训练数据的平均间距值对所有数据进行标准化。我们还将窗口宽度从-200到250 Hounsfield单元的强度标准化。所有的训练超参数都与之前的分割实验相同。