在本练习中,您将实现K-means算法并将其应用于压缩图像。在第二部分中,您将使用主成分分析(PCA)来寻找面部图像的低维表示。
1.聚类(K-means算法的原理实现)
在本练习中,我们将实现K-means聚类,并使用它来压缩图像。我们将从一个简单的2D数据集开始,以了解K-means是如何工作的,然后我们将其应用于图像压缩。
我们将实施和应用K-means到一个简单的二维数据集,以获得一些直观的工作原理。K-means是一个迭代的,无监督的聚类算法,将类似的实例组合成簇。 该算法通过猜测每个簇的初始聚类中心开始,然后重复将实例分配给最近的簇,并重新计算该簇的聚类中心。 我们要实现的第一部分是找到数据中每个实例最接近的聚类中心的函数。
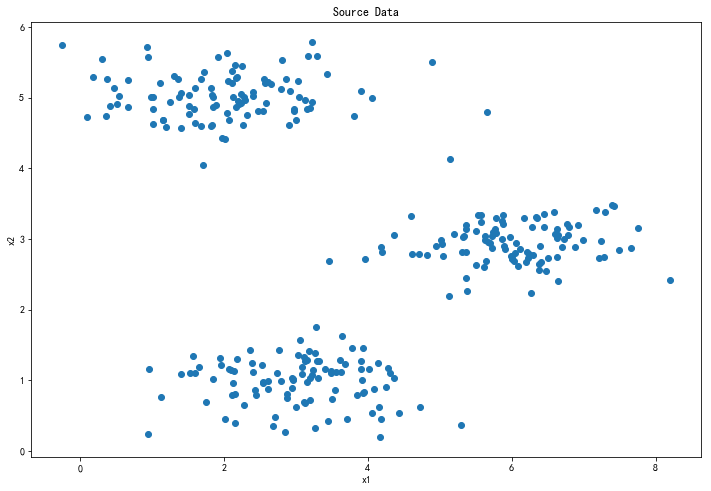
1.1 原始数据
# 展示数据
plt.figure(figsize=(12, 8))
plt.scatter(data['x1'], data['x2'])
plt.title('Source Data')
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()
通过观察可以看出,数据应该被分为3类。
1.2 寻找聚类中心(种子点)
首先要随机初始化种子点,抽取样本中的三个点即可。
# 随机初始化种子点
centroids = np.array(data.sample(3))
计算样本点到每个中心的距离,并找到最小值,确定每个样本所对应的的。

def find_closest_centroids(X, centroids):
"""
找到每个样本距离最近的种子点
"""
# 样本数
m = X.shape[0]
# 分类数
k = centroids.shape[0]
# 储存样本类别的列表
idx = np.zeros(m)
# 迭代寻找最小距离
# 抽取样本
for i in range(m):
# 最小距离,预设为100000
min_dist = 100000
# 抽取种子点
for j in range(k):
# 计算距离
dist = np.sum((X[i,:] - centroids[j,:]) ** 2)
# 判断是否小于前一个存储的距离
if dist < min_dist:
min_dist = dist
idx[i] = j
return idx
返回每个样本所对应的类别的列表。
1.3 绘制聚类结果
def plot_cluster(k, idx, X):
"""
绘制K-means结果
"""
fig, ax = plt.subplots(figsize=(12,8))
for i in range(k):
# 循环绘制每一类
cluster = X[np.where(idx == i)[0], :]
ax.scatter(cluster[:,0], cluster[:,1], s=30, cmap=plt.cm.gist_rainbow, label='Cluster {}'.format(i))
ax.legend()
plt.show()
第一次样本与随机初始化的聚类中心匹配的结果如图:
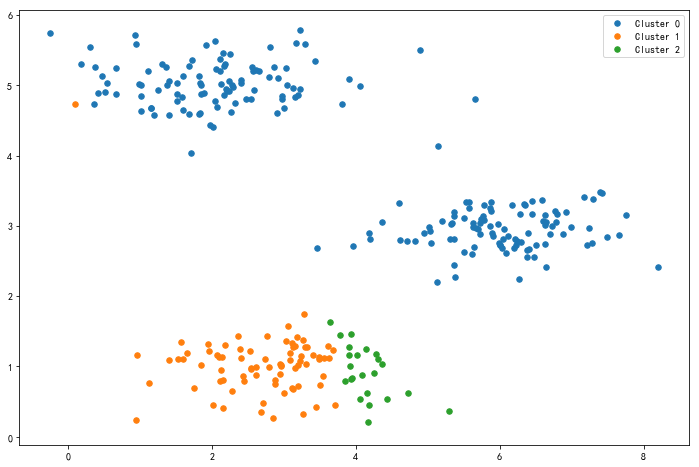
1.4 计算新的聚类中心
接下来,我们需要一个函数来计算簇的聚类中心。 聚类中心是当前分配给簇的所有样本的平均值。
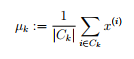
def compute_centroids(X, idx, k):
"""
计算新的中心,k:类别数
"""
# m:样本数,n:特征数
m, n = X.shape
# 初始化存储聚类中心的矩阵
centroids = np.zeros((k, n))
# 循环计算不同类的中心
for i in range(k):
# 返回idx==i位置的索引号,元组类型
samples_id = np.where(idx == i)
# 按列求平均数
centroids[i,:] = (np.sum(X[samples_id[0],:], axis=0) / len(samples_id[0])).ravel()
return centroids
其中np.sum(X[samples_id[0],:], axis=0求出了不同类中每个特征值的平均数。
使用新的聚类中心再次与样本进行匹配,得到的结果如图:
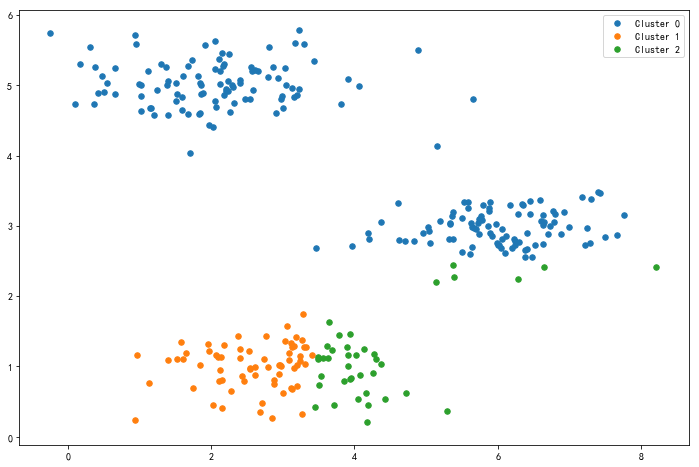
1.5 构建算法模型
综上所述,我们只需要重复这一过程即可:
- 样本和聚类中心匹配
- 计算新的聚类中心
def run_k_means(X, centroids, max_iters):
"""
max_iters:最大迭代次数
"""
# m:样本数,n:特征数
m, n = X.shape
# 类别数
k = centroids.shape[0]
# 迭代
for i in range(max_iters):
# 寻找样本所属类别
idx = find_closest_centroids(X, centroids)
# 计算新的中心点
centroids = compute_centroids(X, idx, k)
return idx, centroids
返回最终的匹配结果和聚类中心,10次循环的结果如图:
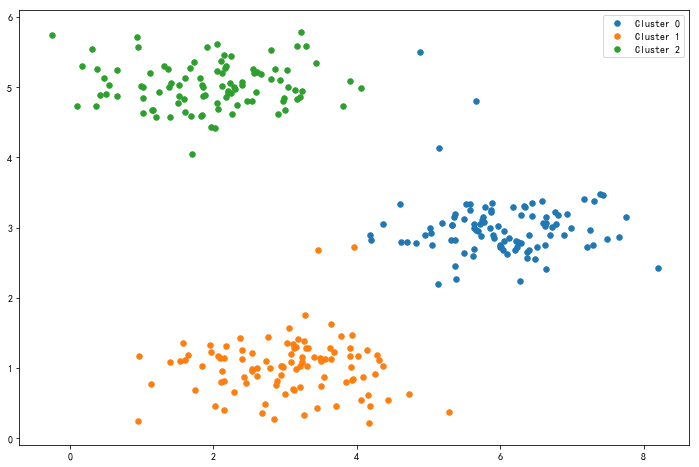
2.K-means用于图像压缩
我们可以使用聚类来找到最具代表性的少数颜色,并使用聚类分配将原始的24位颜色(3 * 8)映射到较低维的颜色空间。
为什么是24位颜色呢?
因为我们所使用的图片(240 * 240)使用RGB空间来表示,有三个图层,而每个图层中的单个像素点都有0-255种状态,故240 * 240的图片中的每个像素点都需要24位的空间表示颜色要素(同时可以理解为有种颜色)。图片所占用的总空间为:240 * 240 * 3 * 8 = 1,382,400 bit。
我们的目标是将原始图片的颜色信息聚为16个类,即用16种颜色来表示,此时颜色要素就只需要4bit空间()。图片所占用的总空间为:240 * 240 * 4 = 230400 bit。与此同时需要额外的空间来存储聚类出的16种颜色,空间为:16 * 3 * 8 = 384 bit。
即将图片压缩了近6倍。
2.1 读取数据
- 原始图片
from IPython.display import Image
Image(filename='data/20180402155901_62188.jpg')
# 读取图片数据
img = plt.imread('data/20180402155901_62188.jpg')
img.shape
形状:(240, 240, 3)

- 数据处理
需要将图片数据由三维转换为二维,由于数据之间相差较大(0-255),故需要特征归一化。
(240, 240, 3) --> (57600, 3)
# 将像素点展开
X = np.reshape(img, (img.shape[0] * img.shape[1], img.shape[2]))
# 特征归一化
X = X / 255
X.shape
2.2 运行K-means算法
# 随机初始化种子点
centroids = np.array(pd.DataFrame(X).sample(16))
# 运行算法
idx, centroids = run_k_means(X, centroids, 10)
聚类结果如图:
这是根据样本中前两行元素的绘图
2.3 观察图片压缩后的结果
接下来需要将原图片中每一个像素点的值替换为它所对应的聚类中心点的值。
# 将每个像素点替换为聚类好的16个类
X_recovered = centroids[idx.astype(int), :]
结果如图:
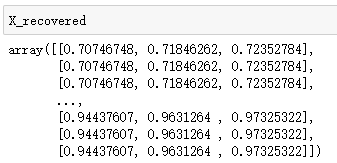
# 将像素点按行列形状组合
X_recovered = np.reshape(X_recovered, (img.shape[0], img.shape[1], img.shape[2]))
plt.imshow(X_recovered)
# 去掉刻度线
plt.xticks([])
plt.yticks([])
plt.show()
可以看到颜色元素变少了。

3.PCA(Principal component analysis)主成分分析
PCA是在数据集中找到“主成分”或最大方差方向的线性变换。它可以用于数据降维。在本练习中,我们首先负责实现PCA并将其应用于一个简单的二维数据集,以了解它是如何工作的。我们从加载和可视化数据集开始。
3.1 加载数据
在进行PCA算法前,我们需要将特征值归一化。使用DataFrame进行归一化是很简单的。
# 特征归一化
X = pd.DataFrame(X)
X = (X - X.mean()) / X.std()
X = np.array(X)
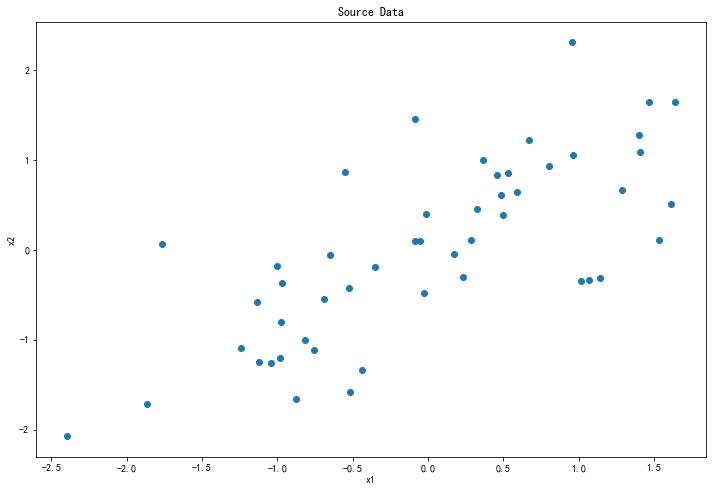
# 数据可视化
plt.figure(figsize=(12, 8))
plt.scatter(X[:, 0], X[:, 1])
plt.title('Source Data')
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()
展示归一化后的原始数据:
3.2 计算协方差矩阵并对其进行奇异值分解
协方差矩阵:
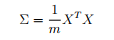
def pca(X):
"""
return: U:特征向量,S:对角矩阵
"""
# 计算协方差矩阵
X = np.matrix(X)
cov = (X.T * X) / X.shape[0]
# 奇异值分解
U, S, V = np.linalg.svd(cov)
return U, S, V
返回的U是一个形状为(n, n)的矩阵,其中n为样本的特征值数量。
3.3 数据降维
现在我们有协方差矩阵的特征向量(矩阵U),我们可以用这些来将原始数据投影到一个较低维的空间中。对于这个任务,我们将实现一个计算投影并且仅选择顶部K个分量的函数,将维数减少到K。
def dimensionality_reduction(X, U, k):
# X的形状(m, n)
# 选择U的K个分量(n, k)
U_reduced = U[:,:k]
# 计算降维后的值(m, k)
Z = np.dot(X, U_reduced)
return Z
返回值Z即为降维后的数据。
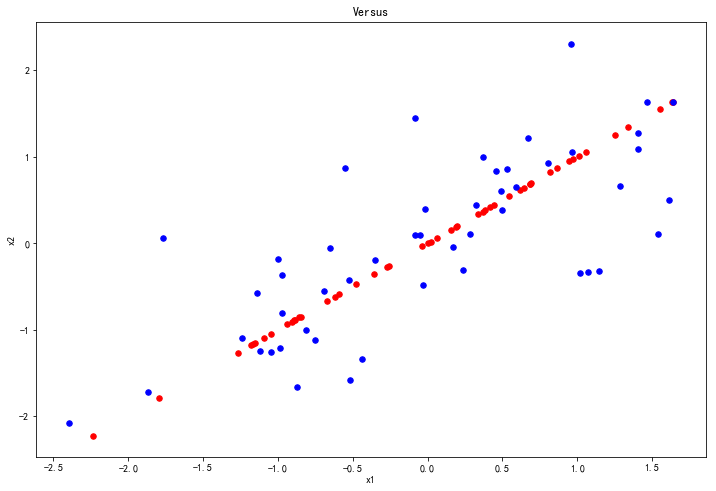
我们来把数据恢复为二维观察一下:
近似的恢复数据,恢复后的数据绘图变成了主方向上的投影。
def recover_data(Z, U, k):
# (n, k)
U_reduced = U[:,:k]
# (m, k) @ (k, n)
X_re = np.dot(Z, U_reduced.T)
return X_re
绘图:
# 数据可视化
fig, ax = plt.subplots(figsize=(12, 8))
ax.scatter(np.array(X_re[:,0]), np.array(X_re[:,1]), s=30, color='r', label='Recover Data')
ax.scatter(X[:, 0], X[:, 1], s=30, color='b', label='Source Data')
plt.title('Versus')
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()
3.4 通过PCA压缩图像
我们在此练习中的最后一个任务是将PCA应用于脸部图像。通过使用相同的降维技术,我们可以使用比原始图像少得多的数据来捕获图像的“本质”。
3.4.1 数据展示
数据是32 * 32的灰度图像,现在我们将前100幅图画出来。
def plot_100figs(X):
"""
画100幅图
"""
# 取前100个索引
sample_list = np.arange(100)
# 从数据中取出这100个值
sample_data = X[sample_list, :]
# 画图, sharex,sharey:子图共享x轴,y轴, ax_array:一个10*10的矩阵
fig, ax_array = plt.subplots(nrows=10, ncols=10, sharex=True, sharey=True, figsize=(8, 8))
# 画子图
for r in range(10):
for c in range(10):
ax_array[r, c].matshow(sample_data[10 * r + c, :].reshape((32, 32)).T,
cmap=plt.cm.gray)
# 去掉刻度线
plt.xticks([])
plt.yticks([])
plt.show()
原始数据:

3.4.2 数据降维
显然我们的每一张图片都有32 * 32 = 1024个特征值。
- 现在我们通过PCA取前100个主成分
U, S, V = pca(X)
Z = dimensionality_reduction(X, U, 100)
X_re100 = recover_data(Z, U, 100)
结果如图:
可以发现图片丢失了细节,但大体轮廓被保留了下来。

- 如果只取10个主成分呢
U, S, V = pca(X)
Z = dimensionality_reduction(X, U, 10)
X_re10 = recover_data(Z, U, 10)
结果如图:

- 如果只取1个主成分呢
U, S, V = pca(X)
Z = dimensionality_reduction(X, U, 1)
X_re1 = recover_data(Z, U, 1)
结果如图:

番外篇
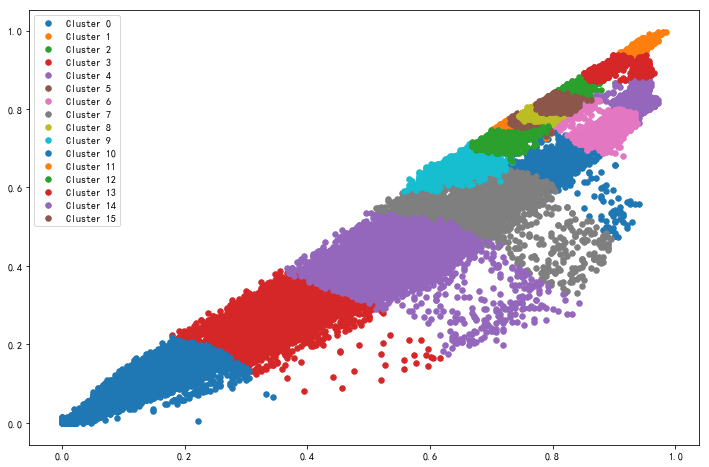
你可以注意到,在使用K-means算法时我们对RGB图像的颜色信息进行了压缩,那么如果使用PCA对颜色信息进行压缩会有什么结果呢?
还是用这张图片:


由此可见,在不同的应用场景使用合适的算法是非常必要的。